Fusing Sequence and Structural Information for Unified Protein
Representation Learning
FusionProt
1 蛋白质表示学习:
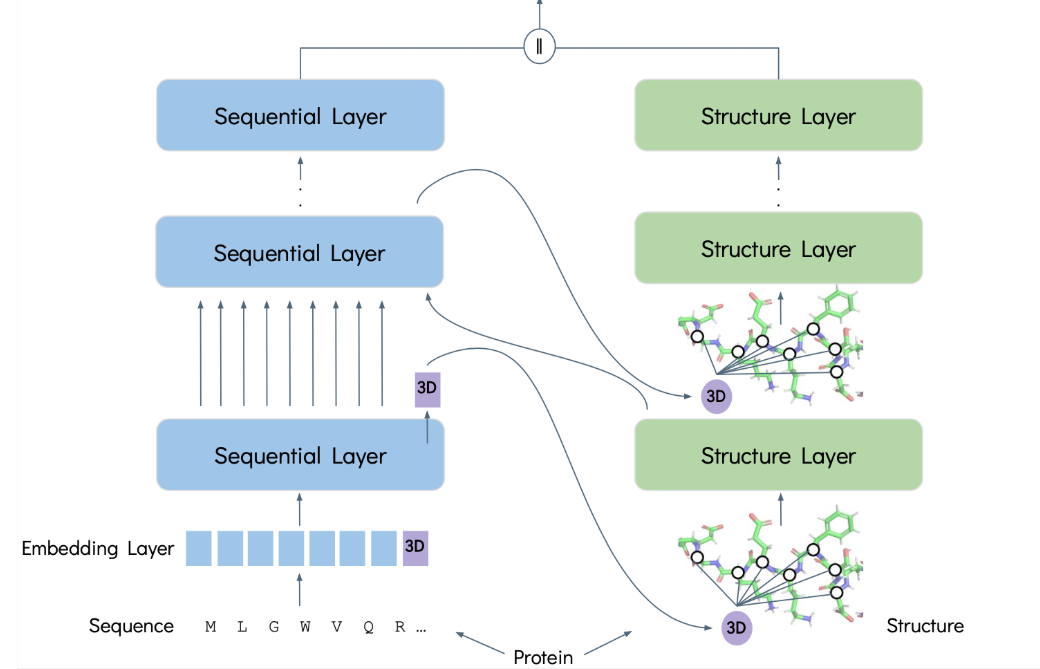
FusionProt :可学习融合
token和迭代双向信息交换,实现序列与结构的动态协同学习,而非静态拼接。
2.
一维(1D)氨基酸序列和三维(3D)空间结构:
好的,完全没有问题。这是对 FusionNetwork
模型架构代码的中文复述分析。
3. 模型总体
1 2 3 4 5 6 7 8 @R.register("models.FusionNetwork" class FusionNetwork (nn.Module, core.Configurable): def __init__ (self, sequence_model, structure_model, fusion="series" , cross_dim=None ): super (FusionNetwork, self ).__init__() self .sequence_model = sequence_model self .structure_model = structure_model self .output_dim = sequence_model.output_dim + structure_model.output_dim self .inject_step = 5
class FusionNetwork(...)nn.Module。__init__(...)sequence_model 和
structure_model 作为输入。self.output_dimself.inject_step = 5每经过序列模型的 5
层,就会进行一次信息交换 。
1 2 3 4 raw_input_dim = 21 self .structure_embed_linear = nn.Linear(raw_input_dim, structure_model.input_dim)self .embedding_batch_norm = nn.BatchNorm1d(structure_model.input_dim)
self.structure_embed_linearself.embedding_batch_norm
1 2 3 4 structure_token = nn.Parameter(torch.Tensor(structure_model.input_dim).unsqueeze(0 )) nn.init.normal_(structure_token, mean=0.0 , std=0.01 ) self .structure_token = nn.Parameter(structure_token.squeeze(0 ))
self.structure_tokennn.Parameter)。这个“令牌”不代表任何真实的原子或氨基酸,而是一个抽象的载体。在训练过程中,它将学习如何编码和表示整个蛋白质的全局
3D 结构信息 。它就像一个信息信使。
1 2 3 self .structure_linears = nn.ModuleList([...])self .seq_linears = nn.ModuleList([...])
self.structure_linears /
self.seq_linears
4. 前向
1 2 3 def forward (self, graph, input , all_loss=None , metric=None ): new_graph = self .build_protein_graph_with_3d_token(graph)
首先调用辅助函数,将输入的蛋白质图谱进行改造:为图谱增加一个代表“3D
令牌”的新节点,并将这个新节点与图中所有其他节点连接起来。
序列模型的初始化 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 sequence_input = self .sequence_model.mapping[graph.residue_type] sequence_input[sequence_input == -1 ] = graph.residue_type[sequence_input == -1 ] size = graph.num_residues if (size > self .sequence_model.max_input_length).any (): starts = size.cumsum(0 ) - size size = size.clamp(max =self .sequence_model.max_input_length) ends = starts + size mask = functional.multi_slice_mask(starts, ends, graph.num_residues) sequence_input = sequence_input[mask] graph = graph.subresidue(mask) size_ext = size if self .sequence_model.alphabet.prepend_bos: bos = torch.ones(graph.batch_size, dtype=torch.long, device=self .sequence_model.device) * self .sequence_model.alphabet.cls_idx sequence_input, size_ext = functional._extend(bos, torch.ones_like(size_ext), sequence_input, size_ext) if self .sequence_model.alphabet.append_eos: eos = torch.ones(graph.batch_size, dtype=torch.long, device=self .sequence_model.device) * self .sequence_model.alphabet.eos_idx sequence_input, size_ext = functional._extend(sequence_input, size_ext, eos, torch.ones_like(size_ext)) tokens = functional.variadic_to_padded(sequence_input, size_ext, value=self .sequence_model.alphabet.padding_idx)[0 ] repr_layers = [self .sequence_model.repr_layer] assert tokens.ndim == 2 padding_mask = tokens.eq(self .sequence_model.model.padding_idx)
序列数据进行 Transformer 模型(如 ESM)所需的标准预处理。
包括添加序列开始(BOS)和结束(EOS)标记,以及将所有序列填充(Padding)到相同长度,以便进行批处理。
模型初始化与初次融合 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30 31 32 33 34 35 36 37 x = self .sequence_model.model.embed_scale * self .sequence_model.model.embed_tokens(tokens) if self .sequence_model.model.token_dropout: x.masked_fill_((tokens == self .sequence_model.model.mask_idx).unsqueeze(-1 ), 0.0 ) mask_ratio_train = 0.15 * 0.8 src_lengths = (~padding_mask).sum (-1 ) mask_ratio_observed = (tokens == self .sequence_model.model.mask_idx).sum (-1 ).to(x.dtype) / src_lengths x = x * (1 - mask_ratio_train) / (1 - mask_ratio_observed)[:, None , None ] structure_hiddens = [] batch_size = graph.batch_size structure_embedding = self .embedding_batch_norm(self .structure_embed_linear(input )) structure_token_batched = self .structure_token.unsqueeze(0 ).expand(batch_size, -1 ) structure_input = torch.cat([structure_embedding.squeeze(1 ), structure_token_batched], dim=0 ) structure_token_expanded = self .structure_token.unsqueeze(0 ).expand(x.size(0 ), -1 ).unsqueeze(1 ) x = torch.cat((x[:, :-1 ], structure_token_expanded, x[:, -1 :]), dim=1 ) padding_mask = torch.cat([padding_mask[:, :-1 ], torch.zeros(padding_mask.size(0 ), 1 ).to(padding_mask), padding_mask[:, -1 :]], dim=1 ) size_ext += 1 if padding_mask is not None : x = x * (1 - padding_mask.unsqueeze(-1 ).type_as(x)) repr_layers = set (repr_layers) hidden_representations = {} if 0 in repr_layers: hidden_representations[0 ] = x x = x.transpose(0 , 1 ) if not padding_mask.any (): padding_mask = None
将 3D 令牌插入序列。
为序列数据生成初始的词嵌入表示 x。
将 self.structure_token 的初始状态插入到序列嵌入
x 中,通常是放在序列结束标记(EOS)之前。
序列模型看到的输入序列变成了
[BOS, 残基1, 残基2, ..., 残基N, **3D令牌**, EOS]
的形式。
融合循环 1 2 3 4 5 6 7 8 for seq_layer_idx, seq_layer in enumerate (self .sequence_model.model.layers): x, attn = seq_layer( x, self_attn_padding_mask=padding_mask, need_head_weights=False , ) if (seq_layer_idx + 1 ) in repr_layers: hidden_representations[seq_layer_idx + 1 ] = x.transpose(0 , 1 )
模型开始逐层遍历序列模型的所有层(例如 Transformer
的编码器层)。x 在每一层都会被更新。
1 if seq_layer_idx > 0 and seq_layer_idx % self .inject_step == 0 :
信息注入点 :每当层数的索引能被
inject_step (即 5) 整除时,就触发一次信息交换。
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 if structure_layer_index == 0 : structure_input = torch.cat((structure_input[:-1 * batch_size], x[-2 , :, :]), dim=0 ) else : structure_input = torch.cat((structure_input[:-1 * batch_size], self .seq_linears[structure_layer_index](x[-2 , :, :])), dim=0 ) hidden = self .structure_model.layers[structure_layer_index](new_graph, structure_input) if self .structure_model.short_cut and hidden.shape == structure_input.shape: hidden = hidden + structure_input if self .structure_model.batch_norm: hidden = self .structure_model.batch_norms[structure_layer_index](hidden) structure_hiddens.append(hidden) structure_input = hidden updated_structure_token = self .structure_linears[...](structure_input[-1 * batch_size:]) x = torch.cat((x[:-2 , :, :], updated_structure_token.unsqueeze(0 ), x[-1 :, :, :]), dim=0 ) structure_layer_index += 1
信息流程 :
从序列到结构 :模型从序列表示 x
中提取出“3D
令牌”的最新向量。这个向量此时已经吸收了前面几层序列模型的上下文信息。然后,通过(seq_linears)将其转换后,更新到结构模型的输入中。结构信息处理 :运行一层结构模型(GNN)。GNN
根据图的连接关系更新所有节点的表示,当然也包括“3D
令牌”这个特殊节点。从结构到序列 :从 GNN 的输出中,再次提取出“3D
令牌”的向量。这个向量包含更新后的结构信息。再通过(structure_linears)转换后,把它插回 到序列表示
x 中,替换掉旧的版本。
这个循环不断重复。
输出 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30 31 32 33 34 35 if self .structure_model.concat_hidden: structure_node_feature = torch.cat(structure_hiddens, dim=-1 )[:-1 * batch_size] else : structure_node_feature = structure_hiddens[-1 ][:-1 * batch_size] structure_graph_feature = self .structure_model.readout(graph, structure_node_feature) x = self .sequence_model.model.emb_layer_norm_after(x) x = x.transpose(0 , 1 ) if (seq_layer_idx + 1 ) in repr_layers: hidden_representations[seq_layer_idx + 1 ] = x x = self .sequence_model.model.lm_head(x) output = {"logits" : x, "representations" : hidden_representations} residue_feature = output["representations" ][self .sequence_model.repr_layer] residue_feature = functional.padded_to_variadic(residue_feature, size_ext) starts = size_ext.cumsum(0 ) - size_ext if self .sequence_model.alphabet.prepend_bos: starts = starts + 1 ends = starts + size mask = functional.multi_slice_mask(starts, ends, len (residue_feature)) residue_feature = residue_feature[mask] graph_feature = self .sequence_model.readout(graph, residue_feature) node_feature = torch.cat(...) graph_feature = torch.cat(...) return {"graph_feature" : graph_feature, "node_feature" : node_feature}
提取输出 :循环结束后,分别从两个模型中提取最终的特征表示。读出(Readout) :使用一个“读出函数”(如求和或平均)将节点级别的特征聚合成一个代表整个蛋白质的图级别特征。最终组合 :将来自序列模型和结构模型的节点特征(node_feature)和图特征(graph_feature)分别拼接(concatenate)起来。返回结果 :返回一个包含组合后特征的字典,可用于下游任务(如功能预测、属性回归等)。